文章题目:Deep MALDI-MS spatial omics guided by quantum cascadelaser mid-infrared imaging microscopy
发表期刊:NatureCommunications
研究单位:德国曼海姆应用科技大学仪器分析与生物分析研究所
研究技术:激光面阵列红外成像、MALDI质谱成像
样本类型:小鼠肾脏、小鼠大脑、小鼠脊髓等

2025年5月22日,由德国曼海姆应用科技大学仪器分析与生物分析研究所的Carsten Hopf教授带领的研究团队在《Nature Communications》上发表了题为Deep MALDI-MS spatial omics guided by quantum cascade laser mid-infrared imaging microscopy的论文。在这项研究中,研究团队采用了布鲁克的激光面阵列红外成像光谱仪HYPERION II ILIM对小鼠肾脏、大脑和脊髓等组织进行了超快速成像,该成像技术可以非标记、无损地快速完成整块切片组织的成像,同时还能达到低于1 µm的亚细胞级空间分辨率,这一技术可以快速锁定切片组织的ROI区域,节省95%以上的质谱数据采集时间。这项技术与质谱成像技术的集合,很好的克服了质谱成像数据采集速度慢的局限。在同样的时间内,可以进行更多样品的深度质谱空间组学研究,为病理医学相关领域的研究注入了新的活力。

1. 激光红外成像快速锁定目标测试区域,告别质谱成像“全组织盲扫"
在空间组学研究中,高置信度的分子鉴定对于复杂生物学机制探索和空间生物标志物发现至关重要。然而,当前基于质谱成像的空间组学技术必须在数据采集速度与生化分析深度之间做出妥协。本研究引入快速、免标记的激光面阵列红外成像技术,引导质谱成像精准定位至微小目标区域,很好的解决了这一矛盾。
从下图可以看出,激光面阵列红外成像技术完成整片鼠脑组织切片的成像仅需10分钟,并且空间分辨率高达5微米,通过对红外光谱进行二阶导运算,找出1466cm−1(CH₂弯曲振动)和1742cm−1(C=O振动)这两个特征峰来区分小脑纤维束(FT)和颗粒层(GL),从而将质谱成像的目标测试区域锁定在一更小区域,成功避免了“全组织盲扫"。以10微米的空间分辨率对确定的目标测试区域进行质谱成像,用时7小时,相比于整片全扫,节省了大量测试时间。
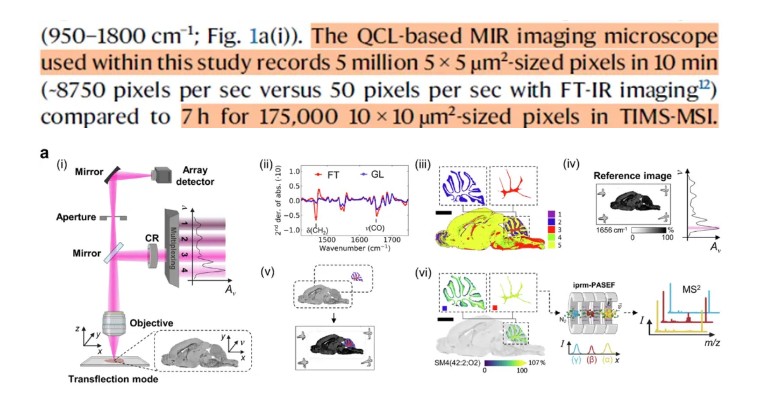
图1.激光面阵列红外成像通过空间上的引导锁定质谱成像的数据采集区域
((i)基于量子级联激光器的红外成像显微镜概述,以及激光红外成像引导质谱成像的具体流程。(ii)通过对光谱吸光度值进行二阶导运算,找出1466cm−1(CH₂弯曲振动)和1742cm−1(C=O振动)这两个特征峰来区分小脑纤维束(FT)和颗粒层(GL)。(iii)基于所选特征峰得到的分布图像和目标测试区域定义。(iv)基于单波数(1656cm-1)得到的参考图像。(v)基于所有高光谱数据集得到的图像与基于1656cm-1单波数得到的参考图像叠加显示。(vi)通过iprm-PASEF对锁定的小脑目标测试区域进行质谱成像。)

2. 激光红外成像快速精准锁定微小测试区域,与耗时的免疫组化结果吻合
在体外3D类癌细胞模型的有氧糖酵解中,QCL-MIR 展现了强大的区分能力:在由成纤维细胞 (CCD-1137Sk) 和阳性全角蛋白克隆癌细胞 (HT-29) 组成的双培养球体中,激光面阵列红外成像技术通过1466 cm⁻¹(CH₂弯曲振动) 和1742 cm⁻¹ (C=O 振动) 这两个脂质相关的红外特征峰的分析,精准区分出成纤维细胞核心与癌细胞外层,与 MALDI 免疫组化结果吻合。后续的质谱成像分析发现:癌细胞中甘油磷脂 (PI 34:1) 含量更高,而成纤维细胞中溶解甘油磷脂(LPI 18:0) 含量更高,揭示了肿瘤微环境中脂质代谢的差异化重塑。
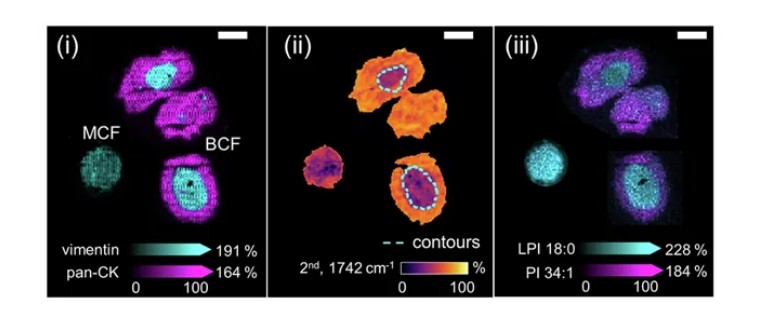
图2.CCD−1137Sk 成纤维细胞/HT-29 癌细胞共培养 (BCF) 体系与单细胞成纤维细胞培养 (MCF) 球体的多模态比较。
((i)使用波形蛋白抗体 (m/z 1230.84; 成纤维细胞) 和全角蛋白抗体 (m/z 1288.71; 癌症) 抗体的多通道MALDI免疫组化质谱成像图 。质量窗口±10ppm。(ii)球体切片的红外成像图 (脂质特征峰1742 cm−1; 二阶导),MALDI免疫组化聚类分析 (k = 2) 得出成纤维细胞核心轮廓 (青色,虚线)。(iii)m/z 835.54 (PI 34:1 [M-H]-; 成纤维细胞) 和m/z 599.32 (lyso-PI 18:0 1 [M-H]-; 癌症) 质谱成像图。比例尺, 200 μm)

3.对肾小球进行测试,进一步验证激光红外成像锁定目标测试区域的准确性
利用酰胺I带和羰基振动之间1720 cm−1处的脂质红外特征峰 (一阶导数) 的差异可以区分肾小球结构和周围的肾皮质。从而可以将质谱成像的数据采集时间减少95% (从整片肾脏组织的216,411个像素点降低至8483个像素点)。随后可以将时间和精力重新聚焦到通过使用iprm-PASEF对锁定的肾小球中的10种神经节苷脂进行非常高置信度的鉴定。

图3. (i) 通过ARSA−/−小鼠肾脏(1720 cm−1; 1阶导数)的激光红外成像图锁定的肾小球图像 (绿色轮廓线) 。(ii)激光红外成像图通过高亮的肾小球引导离子图像
m/z 1151.71 (GM3 34:1;O2[M-H]-;质量窗口±10 ppm) 。比例尺, 300 μm。

4.激光红外成像锁定单个目标细胞,以及在动物实验中的应用实例
最后我们测试了激光红外成像锁定单个目标细胞的可行性。尝试了用于运动神经元疾病研究的冷冻小鼠脊髓切片的灰质,通过激光红外成像成功鉴定了单个含有神经元的细胞。不同的运动神经元位置由磷脂酰肌醇PI 38:4的空间分布特征表征,并由H&E显微照片验证。

图4. (i) 实验性自身免疫性脑瘤小鼠脊髓(1722 cm−1; 一阶导数)与锁定的运动神经元细胞的激光红外图像(蓝线)(ii)激光红外成像引导离子图像m/z 885.549 (PI 38:4[M-H]-;质量窗口±10 ppm),突出显示神经元细胞。比例尺,200 μm。

为了验证激光红外成像在引导带有iprm-PASEF技术的TIMS质谱成进而推动生物医学发现方向的潜力,我们研究了实验性自身免疫性脑脊髓炎(EAE)(一种人类多发性硬化症模型)引起的小鼠脊髓白质在疾病高峰时的动态脂质重塑。通过对1466 cm⁻¹处的脂质特征红外特征峰进行二阶求导分析,快速锁定了小鼠脊髓的白质病变区域,与 H&E 染色的病理结果高度一致,将测量时间缩短了20倍。
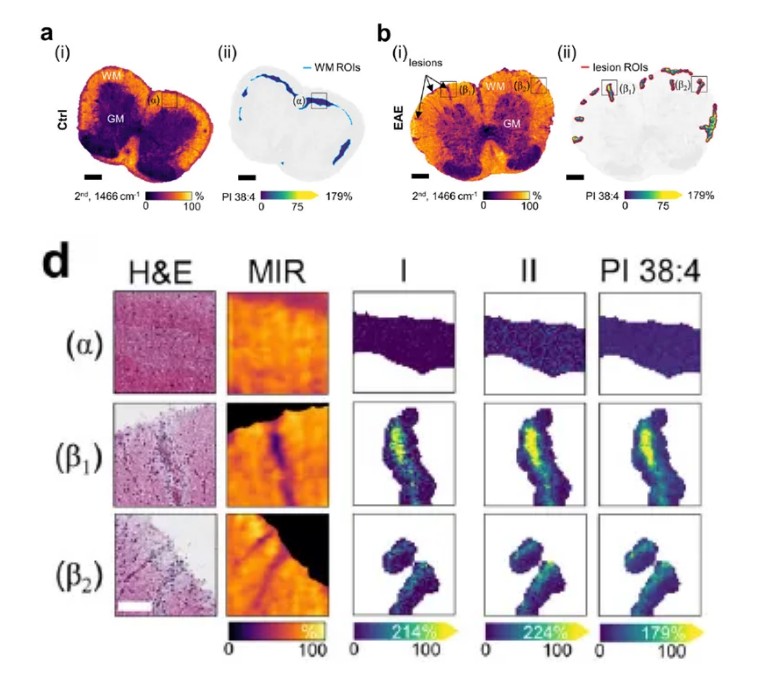
图5.激光红外成像引导 TIMS-MSI 分析实验性自身免疫性脑脊髓炎(EAE)的小鼠脊髓白质中的脂质重塑。a、b (i) 健康对照组的(Ctrl, a)和实验性自身免疫性脑脊髓炎 (EAE, b) 1466 cm−1的小鼠脊髓激光红外图像(二阶导数),脊髓白质中的包含(α -无病变)和(β1),(β2 -变)。比例尺,300 μm。a, b (ii)激光红外成像引导PI 38:4 [M-H]-的质谱成像。相比之下,对照组脊髓(a)的白质比实验性自身免疫性脑脊髓炎(b)的白质区域更小。d 激光红外成像引导离子成像I m/z 616.472 (CerP 34:1;O2[M-H]-) II m/z 687.545 (CerPE 36:1; O2 [M-H]-),和m / z 885.549 (PI 38:4 [M-H]-), 如(a, b)中含有(α),(β1)和(β2)。激光红外图像和H&E显微照片供参考。比例尺, 75 μm。

H&E染色与激光红外成像和质谱成像相结合,提供了疾病高峰期对照组和实验性自身免疫性脊髓炎在脊髓组织学和脂质学变化的多角度视图。I、II和PI 38:4的离子图像显示它们的离子强度显著增加,因此可能在EAE病变区域内积聚。

激光红外显微镜LUMOS II ILIM

联系我们
天津市金贝尔科技有限公司 公司地址:天津市高新区华苑产业园区海泰创新六路华鼎新区一号3号楼10层 技术支持:化工仪器网扫一扫 更多精彩

微信二维码

网站二维码